Hoe zit een eiwit in elkaar? Artificiële intelligentie kan dat voorspellen.
Je staat er misschien niet vaak bij stil, maar je lichaam zit vol met eiwitten, en die voeren allerlei belangrijke functies uit in het lichaam. Zo staan eiwitten in voor het transport van zuurstof doorheen je lichaam, het afbreken van vet of het kunnen opspannen van je spieren.
Vandaag staat eiwitonderzoek meer dan ooit in de belangstelling. Denk alleen al maar aan het onderzoek op de eiwitten van het Coronavirus, dat geleid heeft tot de ontwikkeling van vaccins tegen het virus. Daarenboven zie je technologie steeds meer verwoven worden met biologie. Meer dan ooit tilt artificiële intelligentie biologisch onderzoek naar een hoger niveau, en dat leidde recent tot een echte doorbraak. Maar laten we beginnen bij het begin.
Wat zijn eiwitten en hoe werken ze?
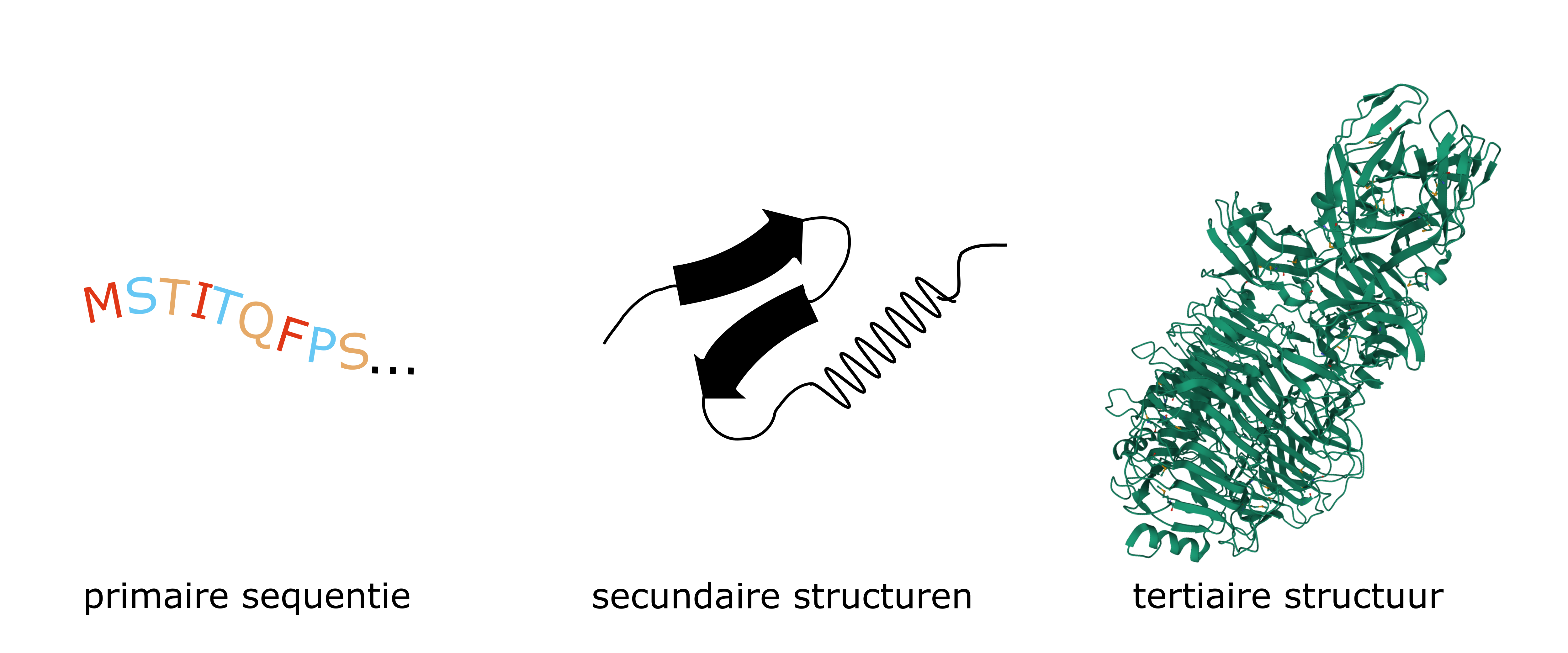
Eiwitten zijn één van de belangrijke klassen macromoleculen die in je lichaam voorkomen. In het lichaam worden ze gevormd startende van de genetische code die in elke menselijke celkern aanwezig is. Die genetische code, het DNA, wordt eerst omgezet tot een gelijkaardige variant (mRNA), waarna het wordt vertaald tot een eiwit (zie ook mijn blog over mRNA vaccins). Elk eiwit is in zijn primaire vorm niets meer dan een ketting van zogenaamde aminozuren (kleine moleculen bestaande uit koolstof, zuurstof, waterstof, stikstof en soms ook zwavel). Die primaire vorm is echter niet hoe eiwitten er in het lichaam finaal uitzien. De primaire vorm vormt lokaal secundaire structuren, en vouwt als het ware in drie dimensies op tot zijn finale en functionele tertiaire structuur. Belangrijk is dat die finale structuur dus afhangt van welke aminozuren er aanwezig zijn en in welke volgorde ze voorkomen. Het is de finale structuur die ook bepaalt welke functie het eiwit in het lichaam kan uitvoeren (in het Engels: form follows function) en net daarom zijn we zo geïnteresseerd in die 3D structuur.
We kennen ondertussen zeer veel van die primaire sequenties, maar slechts van een fractie ervan kennen we ook de echte 3D opvouwing. Dat komt omdat die 3D opvouwing in het labo moeilijk te achterhalen is en ook heel veel kost. Zo kennen we vandaag nog van slechts 17% van alle menselijke eiwitten een experimenteel bepaalde structuur. Om die reden zijn wetenschappers dan ook al decennia lang op zoek naar manieren om die structuren accuraat te kunnen voorspellen, enkel startende van de primaire sequentie. Sinds 1994 is er daarom een tweejaarlijkse internationale competitie, de Critical Assessment of techniques for protein Structure Prediction (CASP), voor onderzoeksgroepen om eiwitstructuren te voorspellen en hun methoden te testen op een objectieve manier.
De verrassing kwam in de voorbije editie van CASP (CASP14). Onderzoekers van de Londonse Google DeepMind zijn er voor het eerst in geslaagd om een score van hoger dan 90% te behalen in de competitie, wat aanzien wordt als de grenswaarde waarboven een voorspelling echt als een correcte oplossing beschouwd kan worden. Sindsdien is DeepMind ook bezig geweest aan het opstellen van een nieuwe database aan voorspelde eiwitstructuren, startende met (onder andere) de menselijke eiwitten. AlphaFold2, de methode van Google DeepMind, maakt gebruik van complexe artificiële intelligentie. En ook andere van de meest accurate methoden (zoals die van Tencent en de Baker Lab groep) maken gebruik van gelijkaardige technieken. Hoe komt het dat artificiële intelligentie ‘plots’ heeft geleid tot een grote doorbraak in het voorspellen van eiwitstructuren?
Artificiële intelligentie en het voorspellen van eiwitstructuren
Yann Lecun, het hoofd van artificiële intelligentie bij Facebook, heeft eens het volgende gezegd over intelligentie:
“Wat ons intelligent maakt is ons vermogen om te denken, begrijpen en leren”.
Het veld van de artificiële intelligentie houdt zich aldus bezig met het bestuderen en ontwikkelen van zowel software als hardware dat kan denken, begrijpen en leren. En vandaag zijn de meest geavanceerde algoritmen hiervoor zogenaamde diepe neurale netwerken. Wiskundig zijn ze eigenlijk niets meer dan een opeenvolgend aantal combinaties en transformaties, maar het zijn net die combinaties en transformaties die (artificiële) neurale netwerken in staat stellen om zeer complexe patronen te herkennen, een taal te leren of zelf met een auto te kunnen rijden (een indrukwekkend voorbeeld is de recente Tesla AI day). Door deze netwerken duizenden tot miljoenen voorbeelden te laten zien van wat ze moeten leren, wordt een zeer complexe wiskundige functie geconstrueerd die iteratief wordt aangepast om beter en beter het onderliggende probleem of fenomeen voor te stellen.
En ook voor het voorpellen van eiwitstructuren is dat het geval. Het doel van het netwerk hier is om zo correct mogelijk de 3D structuur van een eiwit te gaan voorspellen, wat neerkomt op de coördinaten van elk aminozuur in drie dimensies, aan de hand van de primaire sequentie als startpunt. Ook hier leert zo’n netwerk aan de hand van duizenden voorbeelden, of in dit geval sequenties waarvan de structuur al gekend is. Het lijkt op zich redelijk eenvoudig, maar dat is het allerminst. En dat zie je ook aan het grafische overzicht van AlphaFold2 hieronder.
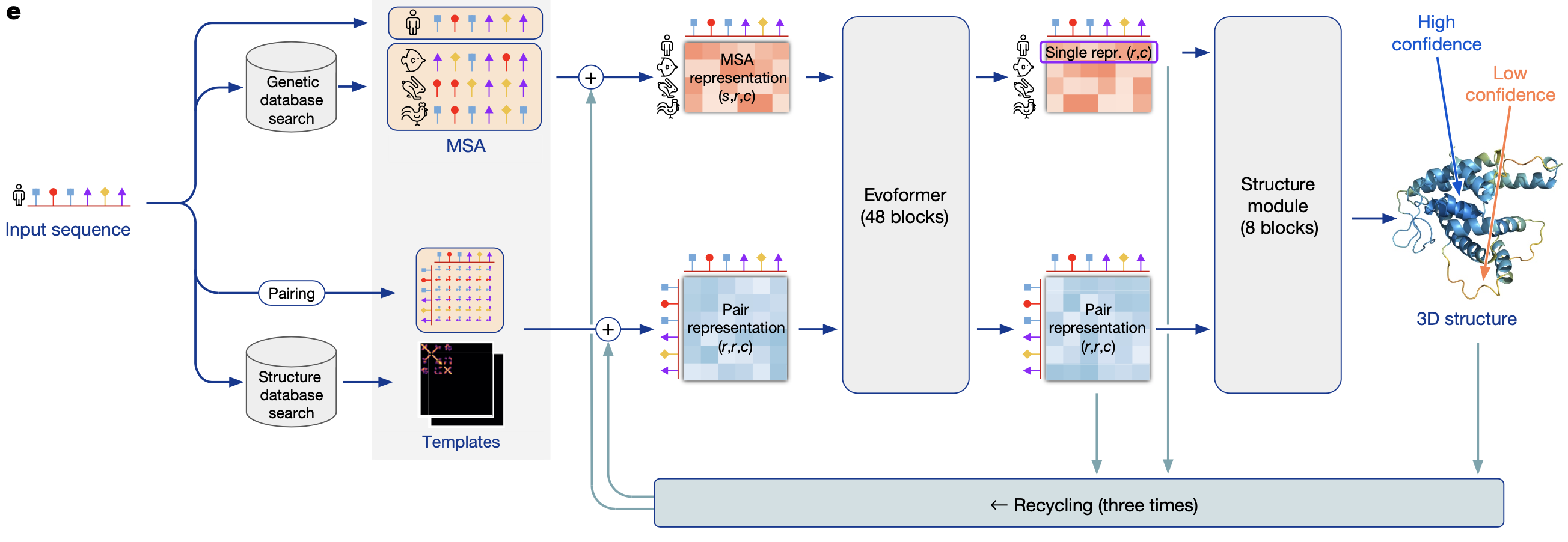
Zonder alle complexe details te overlopen, werkt het AlphaFold2 systeem als volgt. De eiwitsequentie waarvan we de structuur willen voorspellen is het startpunt. Er wordt naar gelijkaardige sequenties gezocht en gekeken of er van die sequenties al 3D structuren gekend zijn (‘templates’). Met de geliijkaardige sequenties wordt dan een multiple sequence alignment (MSA) opgesteld. Dat is een positiegewijze vergelijking tussen alle sequenties om zo gelijkaardige en minder gelijkaardige regio’s in de sequenties te ontdekken. Ten derde worden de templates gebruikt om een pair representation op te stellen, wat een initiële voorspelling voorstelt van welke aminozuren dichtbij elkaar gelegen zijn in drie dimensies.
Het tweede deel van het systeem bestaat uit een zogenaamde Evoformer, wat een zeer specifieke neuraal netwerk architectuur is dat identificeert welke deeltjes van de MSA en pair representation net interessant zijn, en ook informatie tussen deze beide stukken informatie uitwisselt. Finaal worden verfijnde versies van de MSA en pair representation doorgegeven aan het laatste deel van het systeem, de module die effectief de structuur voorspelt. Ook dit deel heeft een complexe netwerk architectuur, waarbij er finaal voor elk aminozuur coördinaten in drie dimensies worden voorspelt. Op die manier is het hele systeem compleet: primaire sequentie in, 3D structuur uit.
Het mooie aan het hele verhaal is dat Google DeepMind hun volledige code ook publiek beschikbaar heeft gemaakt voor iedereen om te gebruiken en verder aan te passen. Helaas lost dat het grootste struikelblok niet op: de immense rekenkracht dat nodig is om die netwerken voorspellingen te laten maken. Andere onderzoekers en onderzoeksgroepen hebben daarom al alternatieven aangeboden. Zo is er RoseTTAFold dat een webserver aanbiedt, en ColabFold die een minder intensieve versie van AlphaFold2 implementeert die ook in de Google Cloud kan lopen. Zulke initiatieven maken het voor veel onderzoekers echt haalbaar om ook praktisch aan de slag te gaan met eiwitstructuren te voorspellen.
Waarom het voorspellen van eiwitstructuren belangrijk is
Maar wat zullen deze nieuwe methoden nu van effect hebben op biologisch onderzoek? Een grote besparing van tijd en geld, dat is zeker. Voorspellingen zullen zeker niet altijd perfect zijn, maar dat maakt deze methoden niet nutteloos. Een voor de hand liggende toepassing is het ontdekken van nieuwe geneesmiddelen. Het kennen van de structuur van een eiwit dat betrokken is in een ziekmakend process is namelijk zeer informatief om er gericht geneesmiddelen tegen te ontwikkelen. Een tweede onderzoeksveld is dat van eiwit design. Net zoals we machines uitvinden om ons te helpen bij bepaalde taken, kunnen we ook eiwitten (nanomachines!) uitvinden om ons te helpen bij verschillende taken. En net omdat de structuur zo belangrijk is voor de functie van een eiwit, kan het design veel sneller verlopen wanneer we accuraat (en veel sneller dan voordien) die 3D structuur te weten kunnen komen. Nog een stapje verder is het begrijpen van eiwit-eiwit interacties, wat ook belangrijk is in veel ziekten.
Kortom, aangezien eiwitten zo belangrijk zijn, en hun structuur zo belangrijk is, brengt het correct voorspellen van die structuren vele mogelijkheden met zich mee!
De afbeeldingen voor deze post zijn (deels) gemaakt door Cuby design van the Noun Project, het RSCB en Jumper et al. (2021).